
今天给大家讲一篇2023年10月在arXiv上发表的 运用深度学习方法进行虚拟筛选 的文章。由于有标签数据的稀疏性,现有深度学习的预测精度无法超过传统对接方法。本文提出了一种新的 对比学习框架DrugCLIP ,无 需依赖于具体的亲和力分数,通过对比学习方法对齐大量成对数据的结合蛋白口袋和分子的表征来根据特定蛋白检索相应的化合物 。此外,实验表明 DrugCLIP在不同的虚拟筛选方法上的性能显著优于传统的对接和有监督学习方法,有助于加速药物发现的进程 。
虚拟筛选技术研究背景
虚拟筛选技术 在计算机辅助药物发现(CADD)中发挥着关键作用,旨在 通过计算方法从庞大的化合物库中寻找与特定蛋白质口袋相互作用的潜在药物分子 。该方法在筛选药物方面的有效性得益于先进的 计算能力 的崛起和大规模 生物分子数据集 的可用性,从而加速了药物发现的进程。
过去,药物化学家们主要关注药物分子与靶标的结合亲和力以及准确的结合位姿。然而,随着化合物库规模的不断增加,这类传统方法的计算成本和筛选效率成为制约因素。然而 虚拟筛选利用计算方法可以快速对大规模的化合物库进行搜索,大大提高了药物发现的效率 。相较于实验室筛选,成本更低,且可以在更短的时间内找到候选药物。
近年来,专家们逐渐将虚拟筛选定义为信息检索任务,即 通过相似性匹配来判断分子与给定蛋白质口袋的相关程度 ,即 从化合物库中筛选出与目标口袋最为相似且有可能结合的分子 。相较于传统的结合亲和力预测或结合位姿的判定的方法,这一方法更注重 潜在结合分子的相似性 ,以提高筛选的精确性和效率。
模型介绍
2.1 DrugCLIP模型架构
DrugCLIP架构 主要分为两个分支,其一为 化合物分支 ,其二为 蛋白 。编码器是一个 SE(3) Transformer ,将每个原子的特征作为输入。为了使得结构保持SE(3)不变性,作者引入了三维坐标的几何距离作为三维特征(图1)。
借鉴了 语言模型bert 的思路,还引入了 随机屏蔽原子类型 方法进行预训练以预测被遮蔽的原子类型。此外,还加入 随机噪声 来打乱原子坐标去重构原始坐标。其中有一个特殊的原子分类标记[CLS]的坐标位于所有原子的中心,该标记用于输出相应蛋白质和分子的表示。损失函数的值为化合物到蛋白口袋的损失(

)加上蛋白口袋到化合物的损失(
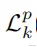
)之和的一半。公式为
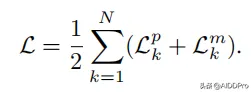
,其中N为化合物蛋白对的数量。
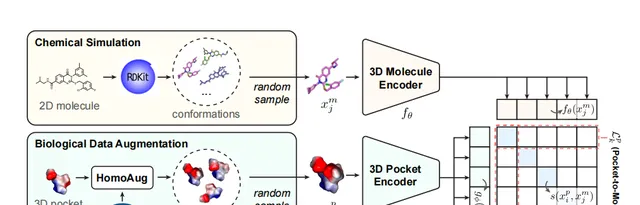
图1DrugCLIP模型架构
2.2 数据增强方法
作者发现在蛋白口袋或分子数据中引入噪声或扰动可能导生成化学上不合法的结构。为了解决这一问题,提出了一种新的 HomoAug增强方法 。它利用生物学中同源蛋白的概念,将PDBbind 的配体与其口袋对应的同源蛋白结合起来,将同源蛋白与配体结合,形成一个增强后的口袋配体对作为新的训练数据(图2)。
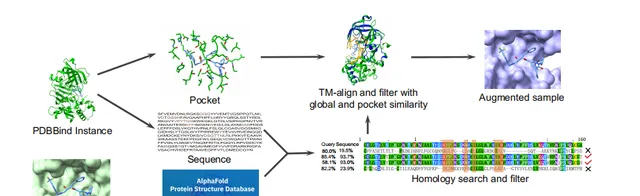
图2 数据增强方法
实验结果
3.1虚拟筛选性能评估
作者分析比较了两种不同的场景:其一是 在不同化合物库上对特定靶标进行一次性虚拟筛选 ,其二是 在特定化合物库中对多个靶标进行多次筛选 。该实验以机器学习打分函数(MLSF)作为基准比较。如图3a所示,当所有的候选分子都没有预先编码时,该方法与其余方法筛选时间相当。然而,如果所有候选分子都进行预先编码,DrugCLIP可以在大致30小时内对包含60亿分子的Enamine数据库进行虚拟筛选。这种时间的显著减少表明了该方法在利用预编码分子时的效率和可伸缩性。当搜索库固定时,所有的分子都预先进行编码。在对10个靶标筛选时,DrugCLIP和MLSF之间的时间差约为10天。这项实验突显了当前基于机器学习的筛选方法的局限性,并表明了DrugCLIP所具有的巨大筛选效率的优势(图3b)。
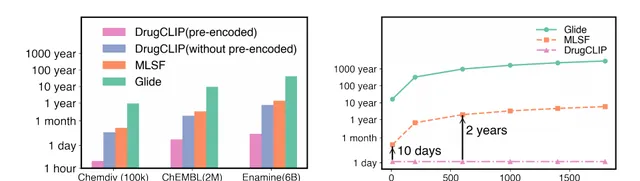
图3 模型预测性能评估
3.2 消融实验评估
作者对两种不同的训练策略进行评估:其一是 使用HomoAug方法进行数据增强 ,其二是 使用RDkit构象来取代原始的分子构象 。如图4所示,在结合两种不同的增强方法时,DrugCLIP模型的分类效果最佳。
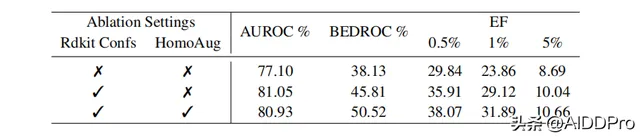
图4消融实验评估
结论
作者提出了一种高效、准确的 虚拟筛选的新方法(DrugCLIP) 。该方法 利用对比学习方法来对齐绑定口袋和分子的表示 。在不同的虚拟筛选任务中该方法均有竞争性的优势,不仅提高了筛选准确性,而且显著缩短了大规模虚拟筛选所需的时间。这为从数十亿种化合物的虚拟库筛选候选药提供了可能性。此外, 通过不断着眼于探索新的数据增强方法及原子级相互作用将助于更有效地发现潜在的候选药物 。
参考文献
- Michael Brocidiacono, Paul Francoeur, Rishal Aggarwal, Konstantin Popov, David Koes, and Alexander Tropsha. Bigbind: Learning from nonstructural data for structure-based virtual screening. 2022
版权信息
本文系AIDD Pro接受的外部投稿,文中所述观点仅代表作者本人观点,不代表AIDD Pro平台,如您发现发布内容有任何版权侵扰或者其他信息错误解读,请及时联系AIDD Pro (请添加微信号sixiali_fox59)进行删改处理。
本文为原创内容,未经授权禁止转载,授权后转载亦需注明出处。有问题可发邮件至[email protected]











