2024年8月28日, 张锋 团队( 徐沛雨 和 Makoto Saito 为论文共同第一作者) 在 Cell 期刊发表了题为:Structural Insights into the Diversity and DNA Cleavage Mechanism of Fanzor 的研究论文【1】 。该研究展示了 Fanzor 蛋白在不同生物中所表现出的分子多样性,并深入解析了其通过RNA引导的DNA切割机制,为进一步的基因工程改造和开发奠定了基础。详情:Cell:张锋团队揭示真核基因编辑系统Fanzor的结构多样性和DNA切割机制
仅仅一天后, 张锋 团队在 Science 期刊发表重磅论文,该研究颠覆了教科书对遗传信息线性传递方式的定义,也颠覆了染色体拥有细胞用来产生蛋白质的一整套完整遗传指令的观点,揭示了「 隐藏基因 」的存在,表明从非编码RNA的基因合成是原核生物中的一种 全新遗传调控方式 。
1958年,DNA双螺旋的发现者之一—— 弗朗西斯·克里克 (Francis Crick) 提出了著名的「 中心法则 」,为遗传信息的传递构建一个大框架: DNA→RNA→蛋白质 。根据这一法则,DNA作为基因模板,转录生成RNA,RNA再翻译为蛋白质。
1970年,科学家们在一些病毒中发现了 逆转录酶 ,这种酶可以将RNA逆转录成DNA,这为中心法则做出了重要补充,揭示了遗传信息并不只是单向流动,也可以实现从RNA→DNA的逆向流动。
自遗传密码被破译以来,我们打开了生命的天书,通过阅读和解码我们的染色体,我们得以识别基因组中的基因,这种 线性的生命法则 被认为支配着所有的生命形式——从细菌到人类。教科书上对 蛋白质编码基因 的定义是它沿着DNA轴线性编码,以便基因的起始端始终位于基因末端的上游。
然而, 张锋 团队的这篇 Science 论文,与哥伦比亚大学 Samuel Sternberg 团队稍早些发表的 Science 论文一起, 颠覆了教科书对蛋白质编码基因的定义 ,揭示了 细菌中特有的抗噬菌体蛋白 ,这些蛋白并非直接由细菌的DNA编码,为了产生这些蛋白,DNA首先被转录为非编码RNA(ncRNA) ,然后通过 滚环逆转录 (rolling circle reverse transcription) 反应将其转换为DNA(即从头产生新基因) ,由此产生的DNA随后被转录为编码抗噬菌体蛋白的mRNA,mRNA再编码为抗噬菌体蛋白,帮助细菌克服病毒(噬菌体) 的感染。


这两项研究 强调了细菌逆转录酶以RNA为模板创造新基因的基因组编码潜力,是对中心法则这一遗传信息传递方式的挑战,也将改变我们看待基因组DNA线性编码遗传信息的传统范式。
逆转录酶 (Reverse transcriptase,RT) 是一种利用RNA模板来合成DNA的酶,广泛分布于生命的所有领域。这些酶在多个过程中发挥作用,包括逆转录病毒和可移动遗传元件的生命周期以及端粒生物学。
在细菌中,逆转录酶对于 抗噬菌体防御 尤为重要,并被多种遗传系统所利用,这些遗传系统的作用是保护细菌免受噬菌体感染。例如,一些 CRISPR-Cas 系统就是利用逆转录来获取对抗RNA噬菌体的新的核酸免疫盒 (spacers) 。逆转录酶也用于被称为 反转录子 (Retron) 的抗噬菌体遗传系统,该系统由三个基因组成,分别编码一个逆转录酶、一个非编码RNA和一个「效应」毒素。通过逆转录过程,反转录子产生一条DNA和RNA共价连接的嵌合核酸链。这种嵌合的DNA-RNA分子的作用尚不清楚,但已知它通过控制效应蛋白的毒性发挥抗噬菌体活性。此外,还有十多个其他细菌防御系统编码逆转录酶,但在大多数这些系统中逆转录的作用尚不清楚。
张锋 团队和 Samuel Sternberg 团队各自独立研究了 肺炎克雷伯菌 ( Klebsiella pneumoniae ) 中的一种包含逆转录酶的防御系统—— 防御相关逆转录酶2 (DRT2) 。
这两项研究均发现,DRT2系统为细菌提供了强大的抗噬菌体防御能力,并且在感染期间,编码DRT2的细菌停止生长和分裂,不允许噬菌体复制。而DRT2系统仅编码一个 逆转录酶 和一个 非编码RNA (ncRNA) ,似乎没有涉及任何其他蛋白质结构域或因子。
因此,这两项研究都进一步探索了这种极简系统究竟是如何既能识别病毒 (噬菌体) 感染又能导致生长停滞的。他们发现,DRT2系统实际上编码了另一种蛋白质,但该蛋白质的产生需要一系列复杂且意料之外的分子事件。
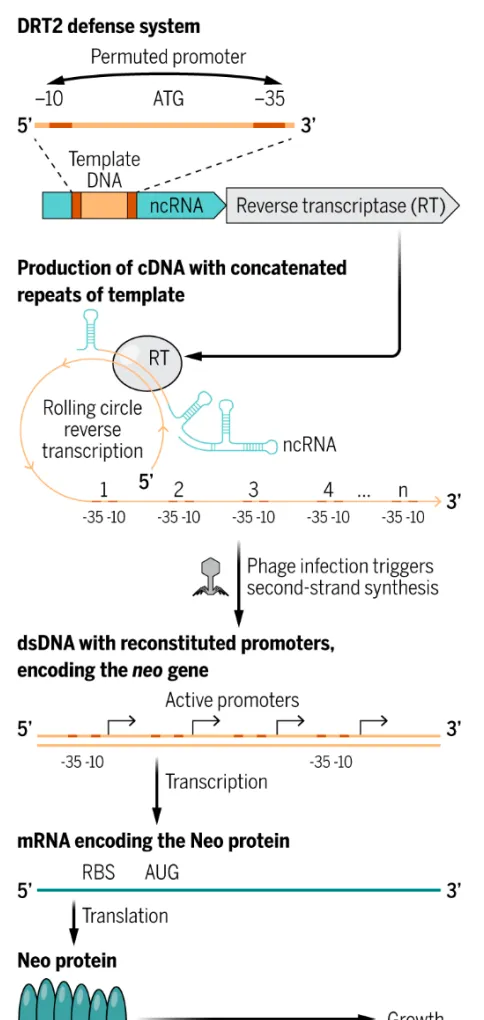
这两项研究观察到,逆转录酶 (RT) 使用相关非编码RNA(ncRNA) 中一段特定的120个碱基作为模板来生成互补DNA(cDNA) 。逆转录酶在一次逆转录过程中不会停止,而是跳回到120个碱基序列的起始点,并以所谓的「滚环」(rolling circle) 反应继续进行,从而生成一个包含多个逆转录模板重复序列的长cDNA。最常见的生成的cDNA包含模板的五个串联重复,有些cDNA甚至包含一百多个这样的重复。
那么, 这些cDNA可能起到什么作用呢?
这两项研究都指出,cDNA包含的序列基序几乎与细菌σ70启动子的共有序列相同。这些启动子由两个序列基序组成,即-35和-10基序 (之所以这样称呼,是因为它们分别位于转录起始位点上游35个和约10个碱基处) 。DRT2系统ncRNA序列中的模板重复包含这两个基序,但排列方式不同,因此单个重复无法形成有效的启动子;但当连接到cDNA上时,一个重复末端的-35基序与下一个重复开头的-10基序处于正确的方向和距离,形成一个强大的启动子,激活了高转录率。
这两项研究进一步确定,该启动子驱动了mRNA转录,而这个mRNA反过来编码了一种蛋白质,从串联的cDNA转录的mRNA不包含任何终止密码子,由于该蛋白质可能会非常长 (取决于cDNA中的重复次数) , 张锋 团队和 Samuel Sternberg 团队都将其命名为 Neo ,意为「 永无止境的开放阅读框 」( N ever- E nding O pen reading frame) ,这一命名也致敬了电影【黑客帝国】中的男主角救世主尼奥(Neo) 。
那么, DRT2系统是如何帮助细菌对抗噬菌体感染的呢?
研究团队发现,DRT2产生单链串联的cDNA,但只有当细菌被噬菌体感染时,第二条链的合成才会启动。一旦形成双链cDNA,启动子就会其驱动转录为mRNA,随后mRN被翻译成Neo蛋白,这种毒蛋白能够迅速抑制细菌生长,从而阻止噬菌体复制和利用细胞资源。
编码Neo蛋白所必需的遗传信息流非常不寻常。这种不常规的蛋白质编码机制有什么好处呢?
研究团队提供了两种可能的解释。 一种解释是 ,有毒基因的表达难以控制,其转录泄漏会导致不良后果,这被认为是编码毒素的防御系统在短的进化时间尺度内往往经常丢失的原因。在DRT2系统中,转录泄漏引起的自身免疫问题得到了解决,因为单个重复序列不包含启动子并且本身无毒。 另一种解释是 ,裂解性噬菌体在感染早期往往会降解宿主的基因组,部分原因是为了关闭细胞通过免疫蛋白的转录和翻译来应对感染的能力。而DRT2系统巧妙地克服了这一挑战,它依赖于一种在感染前已被转录的非编码RNA (ncRNA) ,一旦噬菌体的DNA降解活性停止,就可以将这种ncRNA转换回DNA。
目前尚不清楚DRT2系统如何感知噬菌体的存在,以及是什么分子机制激活了第二条cDNA链的合成。尽管 Samuel Sternberg 团队的研究表明第二条链的合成是从与ncRNA末端共价连接的短DNA引物开始的,但这种引物是如何产生的还不清楚。
此外,还有一个值得关注的问题——Neo蛋白是如何抑制细菌生长的,Neo与任何已知的蛋白质都不相似,它抑制细菌生长的机制仍然是个谜。
来自细菌防御系统的逆转录酶,特别是来自反转录子 (Retron) 的逆转录酶,因其能够生成所选择的cDNA而被用于基因组编辑。DRT2系统通过滚环逆转录生成双链DNA的能力可能有助于生物技术应用,利用其扩增和连接模板序列的能力。此外,细菌防御系统还编码了数十种逆转录酶,而它们的机制尚有待探索。
论文链接 :
1. https://www.cell.com/cell/fulltext/S0092-8674(24)00844-4
2. https://www.science.org/doi/10.1126/science.adq3977
3. https://www.science.org/doi/10.1126/science.adq0876











