染色质免疫沉淀结合测序(ChIP-seq)是一种强大的工具,用于研究体内DNA与组蛋白的相互作用。尽管ChIP-seq已被广泛使用,但在植物中应用仍然具有挑战性,尤其是当样本富含初级或次级代谢物(如种子)或起始材料有限时。传统的ChIP实验由于对大量植物材料的需求以及众多劳动密集型步骤,显著限制了实验的规模,这往往对缺乏ChIP经验的实验室构成障碍。
2024年9月15日, New Phytologist 在线发表了题为 "A simple, robust, cost-effective, and low-input ChIP-seq method for profiling histone modifications and Pol II in plants" 的研究论文。本文发了一种新方法—UP-ChIP(Ultrasensitive Plant Chip-seq),该方法能够减少实验的劳动强度、成本和所需样本量,并且适用于植物中组蛋白修饰和RNA聚合酶II(Pol II)的分析。研究建议使用UP-ChIP替代传统ChIP-seq,尤其是在植物样本中分析组蛋白修饰和Pol II时的起始材料数量有限时。

UP-ChIP方法结合了超低输入的天然ChIP-seq和ChIPmentation的优点,适用于处理多种植物样本(如纯化的细胞核、新鲜幼苗和种子)。其流程(图1)包括使用液氮将样本研磨成粉末,随后通过微球菌核酸酶(MNase)处理释放染色质,一部分碎片作为输入DNA,其余染色质则通过抗体包被的磁珠进行免疫沉淀。沉淀的DNA和输入样本经过Tn5转座酶反应直接添加测序接头,纯化后通过PCR扩增构建文库,并在Illumina平台上进行测序。整个过程可以在2天内完成,并具有高灵敏度、低试剂成本和低抗体需求,相较于传统ChIP方法具有显著优势。
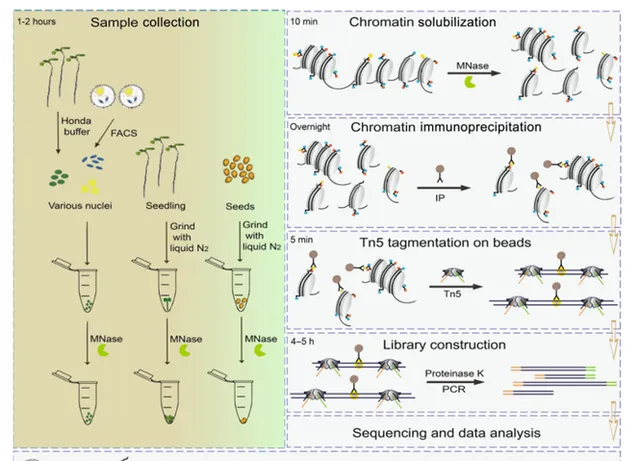
图1 UP-ChIP方法的工作流程
研究中展示了通过UP-ChIP方法在不同质量的拟南芥幼苗细胞核样本(20 mg、10 mg、5 mg和2.5 mg)中对pK27me3组蛋白修饰进行分析的结果(图2)。实验表明,通过Honda缓冲液分离的细胞核样本非常适合UP-ChIP方法。数据质量高,表现为明显的峰值与背景信号差异、大量重叠的峰值、以及符合预期的pK27me3信号覆盖整个基因的模式。即使使用最少量的2.5 mg细胞核样本,实验依然产生了良好的结果,表明UP-ChIP方法对低输入样本具有良好的适用性,并且可能在更低的起始材料下也能获得可接受的结果。
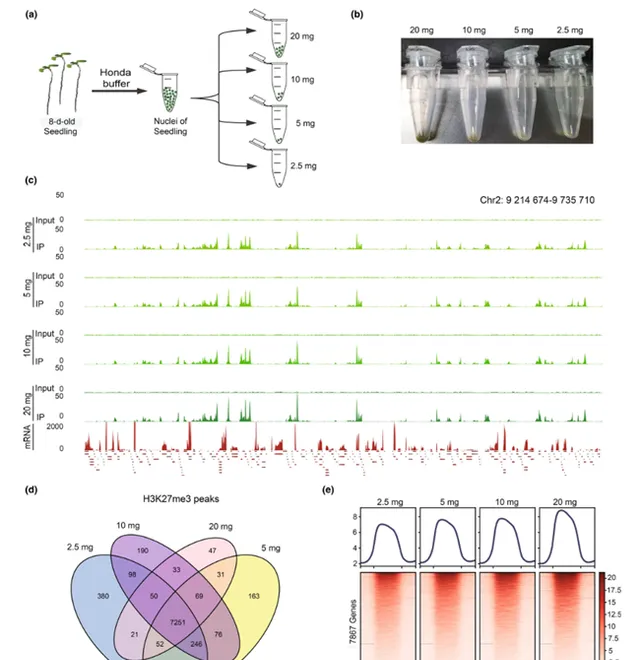
图2 UP-ChIP在不同质量的幼苗核上的灵敏度和可靠性测试
该实验还考虑了简化UP-ChIP流程的可能性,通过直接使用新鲜植物组织而跳过细胞核提取步骤。研究人员对单个拟南芥幼苗及不同数量的种子(20、30、40、50颗)进行了pK27me3 UP-ChIP分析(图3)。结果表明,UP-ChIP在未提取细胞核的情况下,依然能够获得与传统ChIP方法相当的结果,且只需使用少量样本,如单个幼苗或2.5 mg细胞核。对种子的UP-ChIP虽然挑战更大,但通过添加氯仿处理染色质以去除脂肪酸,成功解决了脂肪酸和多糖含量高的问题。实验还发现,通过增加种子数量,可以提高测序比对率。MNase浓度的实验进一步表明,适量的MNase(1-2 U/µl)能产生最佳的ChIP结果,而过高的浓度则会影响信号的连续性和样本的重复性。总体而言,UP-ChIP在植物种子和幼苗中表现出良好的适用性,但对于富含淀粉的种子(如水稻)可能需要进一步优化。
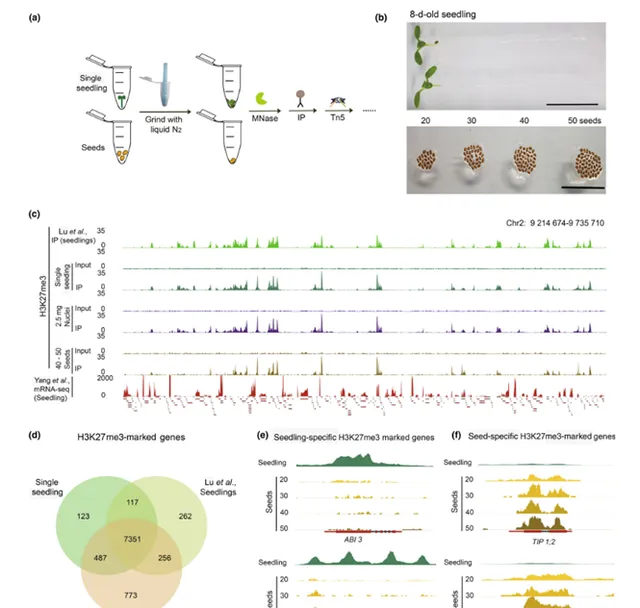
图3 单个幼苗或种子样品的UP-ChIP测试
此外,本研究还展示了UP-ChIP方法在拟南芥幼苗中用于分析RNA聚合酶II(Pol II)结合模式的结果(图4)。与大多数非组蛋白染色质蛋白不同,Pol II在MNase消化中与DNA形成稳定复合体,能够抵抗消化。在实验中,使用针对Pol II Ser2P CTD的抗体进行了UP-ChIP,结果检测到大量Pol II峰,尤其集中在活跃转录的基因3'末端。UP-ChIP获得的Pol II结合图谱与pNET-seq方法的结果高度一致。进一步分析还表明,Pol II和pK27me3的信号呈现相互排斥的模式,验证了UP-ChIP在Pol II分析中的可靠性。总的来说,UP-ChIP可以在不进行化学固定和细胞核提取的情况下,成功分析基因组范围内的组蛋白修饰和Pol II结合,简化了实验步骤。
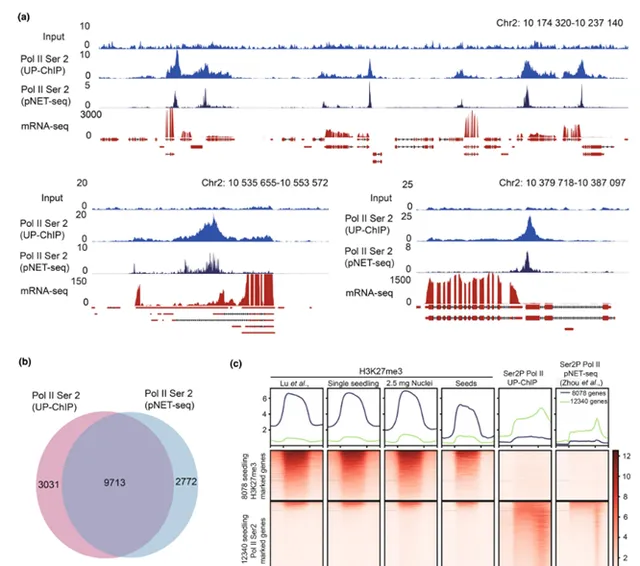
图4 UP-ChIP在拟南芥幼苗中进行Pol II分析的测试
最后,研究中人员开展了UP-ChIP方法在检测拟南芥pK27me3缺失突变体(swn clf)的可行性实验(图5)。PRC2复合体的关键亚基CLF和SWN负责在植物营养组织中沉积pK27me3,抑制基因表达。突变体swn clf中pK27me3的缺失导致幼苗发育缺陷。UP-ChIP对该突变体的分析显示,仅产生背景信号,表明UP-ChIP能够准确反映pK27me3的缺失。进一步的实验通过混合不同比例的野生型Col-0和swn clf提取物,验证了UP-ChIP对组蛋白修饰水平变化的敏感性,结果显示随着swn clf比例的增加,pK27me3信号逐渐减弱。尽管UP-ChIP对组蛋白修饰变化具有灵敏性,但与其他ChIP-seq方法类似,精确量化仍具有挑战性。
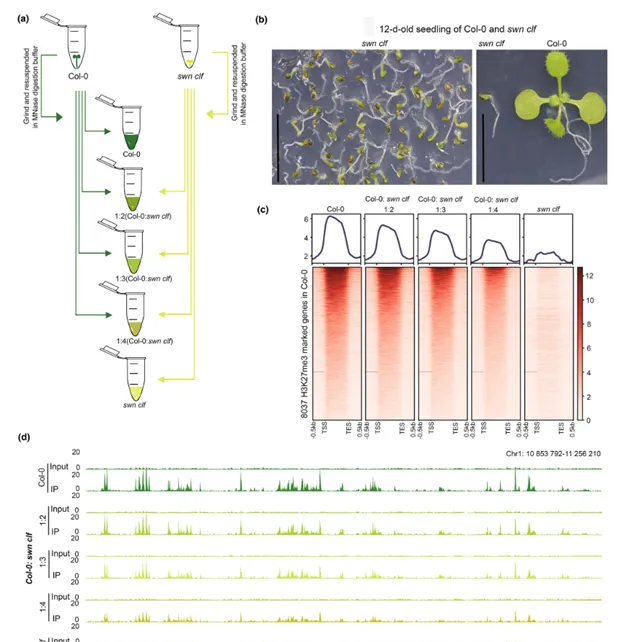
图5 对具有pK27me3的幼苗样本进行UP-ChIP检测
总的而言,UP-ChIP方法是传统ChIP-seq的替代方案,特别适用于样本量有限的情况。它简化了实验流程,省去了繁琐的步骤,同时降低了成本。然而,该方法可能不适用于分析瞬时结合的蛋白质,也难以进行单细胞分析。但是,对于没有丰富资源或ChIP-seq经验的实验室来说,UP-ChIP提供了一种低成本且高效的解决方案,能够在低输入的植物样本中分析表观遗传修饰。这将使得更多的植物遗传学和表观遗传学研究得以实现。
原文链接:
https://doi.org/10.1111/nph.20125
小麦族多组学网站: http://wheatomics.sdau.edu.cn
投稿、合作等邮箱: [email protected]












