點選上方藍字·關註我們
文章標題:A specific phosphorylation- dependent conformational switch in SARS- CoV-2 nucleocapsid protein inhibits RNA binding
文章標題:SARS- CoV- 2核衣殼蛋白中特異性磷酸化依賴的構象開關抑制RNA結合
文章作者:Maiia Botova, Aldo R. Camacho- Zarco, Jacqueline Tognetti, Luiza Mamigonian Bessa, Serafima Guseva§, Emmi Mikkola, Nicola Salvi, Damien Maurin, Torsten Herrmann, Martin Blackledge
文章連結:Botova et al., Sci. Adv. 10, eaax2323 (2024) 2 August 2024
雜誌期刊:Science AdvAnceS
實驗背景
本文研究背景:研究了新冠病毒核衣殼蛋白的磷酸化對RNA結合的影響。
對相關研究工作的簡述及評價(分點列出):使用核磁共振技術研究了磷酸化對蛋白結構的影響;發現磷酸化後蛋白與RNA結合被完全抑制;特定激酶的磷酸化模式具有特異性。
本文創新動機:探究特定宿主激酶的招募對調控病毒功能的機制。
實驗方法
本文研究了SARS-CoV-2核衣殼蛋白的磷酸化對RNA結合的影響。透過核磁共振技術,研究了由絲胺酸精胺酸蛋白激酶1、糖原合成酶激酶3和酪蛋白激酶1引起的超磷酸化過程中發生的結構變化,導致與RNA的交互作用被抑制。當八個磷酸化位點均勻分布時,SR區域與單鏈RNA結合相同的界面,完全抑制了RNA結合。這些結果揭示了特定宿主激酶的招募對調控病毒功能的重要性。
實驗結果
本文實驗結果概括:
相關數據集、指標定義、實驗結果:
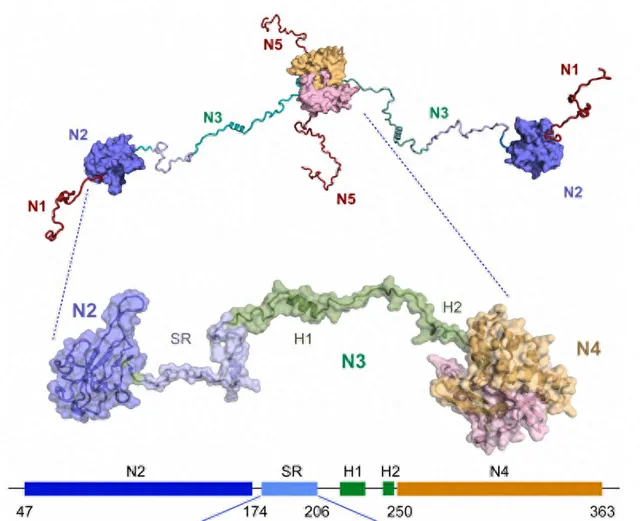
圖示SARS- CoV- 2核衣殼蛋白(N)。SARS- CoV- 2核衣殼蛋白包括5個結構域(n1至n5,頂部)。放大圖中顯示的結構包括三個結構域,n2是RnA結合結構域;n3,包含兩個螺旋p(219 - 231)和p(248 - 255)的無序中心結構域,以及在感染細胞中過度磷酸化的富含絲胺酸-精胺酸(SR)的區域;二聚化區域n4。這個構造稱為n234。SR區序列用藍色表示。

SRPK1、GSK- 3和CK1對N的磷酸化作用。
(A) SRPK1和磷酸化緩沖液[簡稱pn234(i)]孵育後15n- 1h未磷酸化(藍色)和磷酸化(紅色)n234的hSQc比較。兩個共振似乎被分配給pS188和pS206。
(B) pn234(i)與GSK- 3和磷酸化緩沖液[簡稱pn234(ii)]孵育後15n- 1h未磷酸化(藍色)和磷酸化(紅色)n234的hSQc比較。另外出現了8個共振(此處顯示了7個),它們被分配給pS176、pS180、pS184、pS186、pS190、pS194、pt198和pS202。
(C)將未磷酸化(藍色)和磷酸化(紅色)的n234(ii)與cK1和磷酸化緩沖液(簡稱pn234(iii))孵育15n- 1h後hSQc的比較。另外出現了四個共振(圖中顯示了三個),它們被分配給pS193、pS197和pS201。在記錄這些光譜之前,將所有光譜緩沖交換到標準的核磁共振緩沖液中。
(D至F)在所選的共振中,峰強度隨時間的變化,這些共振在整個動力學序列中保持足夠的分辨,以準確測量每一步的峰強度(在磷酸化緩沖液中)。
(D) pn234(i), (E) pn234(ii), (F) pn234(iii)。(G)磷酸化改變了n的動力學行為。主圖:n234在850 Mhz時的旋轉框架弛豫(R1ρ)是磷酸化狀態的函式[藍色,未磷酸化;橙色,pn234 (ii);紅色,pn234 (iii)]。由於低訊雜比,n4的數據未顯示。插圖:950 Mhz下n123的旋轉框架弛豫(R1ρ)[藍色,非磷酸化的n123;橙色,pn123 (ii)]。螺旋p在n123中可見,可能是由於這種結構的分子量較低,可以比較n3的這個區域的動態行為。這一比較表明,過度磷酸化強烈影響SR區域的動態特性。

過度磷酸化模擬RNA與N2的結合。
(A)添加單鏈14- mer RnA (UcUAAAcGAAcUUU)(藍色)後在n234的15n- 1h tROSY光譜中測量的cSPs,與未磷酸化和SRPK1/GSK- 3/cK1磷酸化的n234(紅色)之間的化學位移差異進行比較(由於動態範圍的原因,未顯示由於感興趣的殘基的直接磷酸化而從pSR區域產生的極端位移)。為了與磷酸化cSPs進行最佳比較,將RnA結合的cSPs按因子1.5縮放。
(B)最大化學位移差異對映到n2和n3(黃色)上,以帶狀和表面表示。
(C) RnA結合指和基板β片在RnA結合域上的位置,n2。黃色表面如(B)所示。
(D)在14- mer RnA濃度增加的情況下,由150 μM n234的13c- 1h hMQc得到的化學位移滴定的例子。圖中顯示了RnA結合指(Met101)和基板β片(leu56)上的殘基。這些滴定得到的關聯常數為(230±10)μM。
(E) n234(濃度,150 μM) 15n- 1h tROSY光譜中的cSPs隨14- mer單鏈RnA濃度(紅色,0%;橙色,25%;淺藍色,100%;深藍色,200%)(全光譜見圖S8)。
(F)與(e)相同殘基的n234(濃度,150 μM)在15n- 1h的tROSY光譜中的cSPs與磷酸化狀態的函式關系[紅色,未磷酸化的n234;淺藍色,pn234(i);橙色,pn234 (ii)]。
(G) n234(濃度,150 μM)的13c- 1h hMQc光譜中選定殘基的cSPs隨14- mer單鏈RnA濃度的變化(紅色,0%;橙色,25%;淺藍色,100%;深藍色,200%)。(H) n234(濃度,150 μM)的13c- 1h hMQc光譜中與(e)相同殘基的cSPs作為磷酸化狀態的函式[紅色,未磷酸化的n234;淺藍色,pn234(i);橙色,pn234 (ii)]。

SRPK1、GSK- 3和CK1的過度磷酸化抑制RNA結合。
(A) n234(濃度為150 μM)的13c- 1h hMQc光譜,隨著14- mer單鏈RnA(紅色,0%;深藍色,200%)。
(B) SRPK1/GSK- 3磷酸化的n234 [150 μM pn234(ii)]在13c- 1h hMQc光譜中,選定殘基隨14- mer單鏈RnA濃度的變化而發生的化學位移(紅色,0%;深藍色,200%)。沒有觀察到明顯的變化[與(A)相比]。200%的光譜呈現為單一輪廓,以突出缺乏繫結。
(C)經PKA (150 μM)磷酸化的n234的13c- 1h hMQc光譜中選定殘基的化學位移隨14- mer單鏈RnA濃度的變化(紅色,0%;深藍色,200%)。位移與在未磷酸化的n234中測量的csp具有高度可比性[與(A)相比]。
(D)非磷酸化n123(濃度,200 μM)在存在和不存在等莫耳14- mer單鏈RnA(紅色,0%;藍色,100%)。
(E) SRPK1- GSK3磷酸化n123(濃度,200 μM)在存在和不存在等莫耳14- mer單鏈RnA(紅色,0%;藍色,100%)。100%的光譜呈現為單一輪廓,以突出缺乏結合。
(F) 15n- 1h被PKA磷酸化的n234的tROSY中的cSPs(藍色)和(紅色)pn234(ii)。
(G)未磷酸化的n234(灰色)的13c- 1h hMQc譜圖,n234被PKA(藍色)和pn234(ii)磷酸化(紅色)。
(H)與PKA(灰色/黑色)、SRPK1(綠色)、GSK- 3(藍色)和cK1(紅色)孵育後SR區顯示體外磷酸化位點(黑體)的序列。
(I) n2表面pSR n-末端(182 ~ 191)和c-末端(194 ~ 206)區域的推定差異結合位點示意圖。n2上的紅色陰影表示PKA和SRPK1/GSK- 3磷酸化同時誘導的cSPs,而橙色表示SRPK1/GSK- 3磷酸化僅移位的區域。
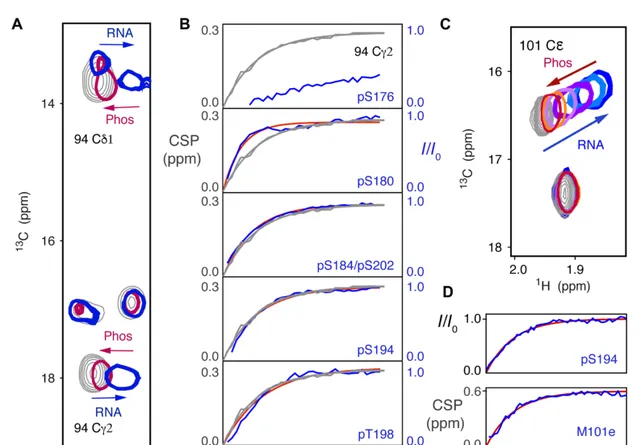
SR磷酸化抑制RNA結合的即時觀察。
(A) pn234甲基13c-1h hMQc中的cSPs(i),特別是94cγ2,顯示了14-mer RnA結合(灰色,0μM,藍色200μM)的影響,以及隨後在與GSK- 3孵育期間相同峰向未結合形式的偏移(紅色)。
(B) 15n-1h hSQc磷酸化峰強度增加的時間過程對比(藍色,實驗;紅色,單指數擬合)和94cγ2的cSP(灰色,實驗和擬合)。94c-γ-2和94c-δ-1的相關時間常數分別為(0.50±0.07)h(pS180)、(0.30±0.04)h(pS184/pS202)、(0.26±0.07)h (pS194)和(0.24±0.05)h (pt198),而94c-γ-2和94c-δ-1的相關時間常數分別為(0.24±0.03)和(0.26±0.03)h。pS176和pS186(未顯示)速度太慢,無法準確擬合。
(C) pn234的甲基13c- 1h hMQc中的cSPs (i) (M101cε)顯示了30- mer polyA RnA結合的影響(灰色,0 μM,藍色120 μM),以及隨後在GSK- 3孵育期間不同磷酸化狀態的分布向未結合的完全磷酸化形式轉移(淺藍色到紅色)。
(D) 15n- 1h hSQc pS194峰強度增加的時間過程(藍色,實驗;紅色,擬合單一指數)和(底部)在與GSK- 3孵育期間,101cε的甲基13c- 1h cSP從RnA結合形式轉變為非結合形式,如圖(c)所示(藍色,實驗;紅色,單指數擬合)。相關時間常數:pS194為(0.10±0.04)小時,101cε為(0.099±0.030)小時。

過度磷酸化對順磁弛豫增強的調節。
(A) 13c- 1h hMQc在teMPO-標記的n234 (G175c)的氧化(藍色)和還原(紅色)形式下的比較。(B) 13c- 1h hMQc在teMPO-標記的pn234(ii) (G175c)的氧化(藍色)和還原(紅色)形式下的比較。在(A)(282和336)中突出顯示的強域間PRes由於磷酸化而明顯減弱。
(C)表明n234中對長程順序的選定影響的表面表示。紅色:n4二聚體疏水表面上的殘基(這兩個單體分別是粉紅色和米色),在teMPO標簽存在下變寬(175),這種交互作用在磷酸化後減弱。黃色:n2上因過度磷酸化而移位的殘基。teMPO標簽的位置以洋紅色顯示。
(D和E)蛋白質的順磁性和反磁性形式之間的強度比。藍色,免費n234;紅色,pn234 (ii)。虛線表示teMPO標簽的位置(175和210)。
(F和G)在自由、非磷酸化形式(A)中,PRes揭示了n2和n4之間以及p和n2之間的長期交互作用。在過度磷酸化後,交互作用被削弱(B)或完全抑制。n2上的csp(黃色)[見(c)]。
實驗結論
本文的貢獻點在於揭示了SARS-CoV-2核衣殼蛋白的一種特定的磷酸化依賴性構象轉換,該轉換會抑制RNA結合。作者使用核磁共振技術研究了三種激酶對核衣殼蛋白的磷酸化作用,並行現當八個磷酸化位點被磷酸化後,核衣殼蛋白的SR區域會與單鏈RNA結合相同的界面,從而完全抑制RNA結合。此外,作者還發現RNA結合、連線和二聚化域之間的遠端聯系被取消,這可能與基因組的包裝和解包裝有關。本文的局限性在於研究僅限於體外實驗,需要進一步的研究來驗證這些發現在細胞內是否成立。總之,本文的研究為了解宿主激酶調節病毒功能提供了新的見解。











