文|vb動脈網
美東時間5月8日,谷歌DeepMind與 Isomorphic Labs(DeepMind 創始人創立)宣布推出新一代AI生物分子結構模型AlphaFlod 3。
據悉,新的模型不僅局限於蛋白質結構的預測,它還能夠預測DNA、RNA、配體等生命分子的結構和相互作用,甚至可以預測轉譯後修飾(PTM)和離子對相應分子系統結構的影響。研究人員僅需輸入一個生物分子復合體的基本描述,幾秒後便能收獲該復合體3D結構的準確預測。
被【Nature 】收錄的【Accurate structure prediction of biomolecular interactions with AlphaFlod 3】 對模型的能力進行了詳細論證。
據論文數據顯示:與現有的預測方法相比,AlphaFlod 3無需輸入任何結構資訊的情況下,其準確性已比PoseBusters基準上的最佳傳統方法高出50%(一些特殊場景可達100%),理論上優於現有的基於物理的生物分子結構預測工具。
不過,任何工具的使用都不能脫離實際。經歷數天測試,已有不少專家學者引入實際問題對AlphaFlod 3的能力評估。就目前測試結果而言,AlphaFlod 3確實充滿潛力,但還不足以「顛覆」這一領域。
全生命分子預測,AlphaFlod 3更接近AIDD了
與過往的AlphaFlod系列工具類似,AlphaFlod 3也采取了神經網絡架構,並以蛋白質數據庫(PDB)中的全球分子結構數據為基礎進行訓練。不過,AlphaFlod 3的預測準度在大部份場景中都遠超前代產品,且在預測範疇上實作了大規模的擴充。
這些能力的升級來源於AlphaFlod 3新引入的主要元件,包括升級版的Evoformer模組(現為Pairformer 模組)、全新的Diffusion Network等。其中,Diffusion Network從點雲透過概率擴散預測座標,進而實作了更高的預測精度。
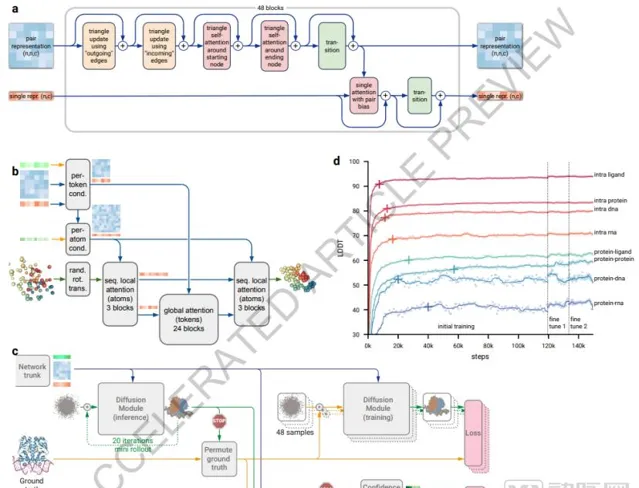
此外,一些模型上的創新也對AlphaFlod 3的預測結果進行了最佳化。在手性分子等形態相似結構上,演算法常會發生預測錯誤。這類情況下,AlphaFlod 3采用了交叉蒸餾的方式,讓具備Transform模型的AlphaFlod 2先行預測,再把預測數據添加到 AlphaFlod 3 的訓練中,一定程度提升了預測的準確率。
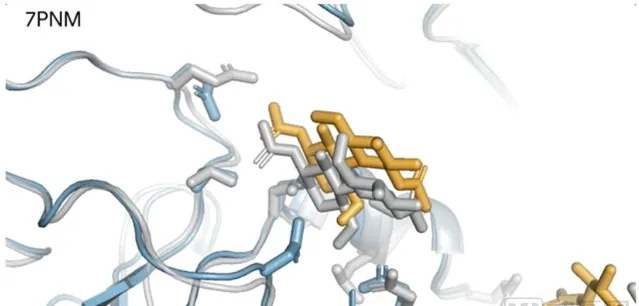
論文展示了部份AlphaFlod 3的預測結果。例如對感冒病毒棘蛋白( 藍色 )與抗體( 綠松石色 )和單糖( 黃色 )相互作用時的結構預測,與真實結構準確匹配( 灰色的)中,它與實驗室得到的結果幾近完全匹配(灰色部份)。
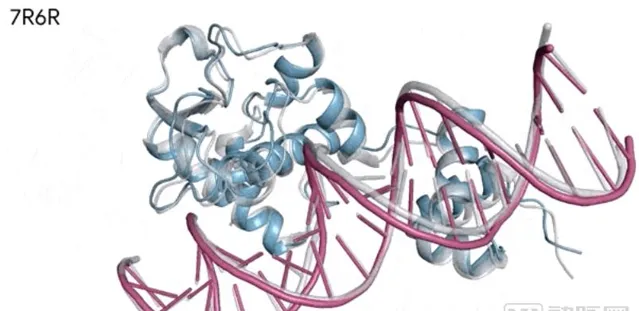
對蛋白質和DNA結合的分子復合物(7R6R - DNA 結合蛋白)進行預測,預測模型也與實驗測定的真實分子結構( 灰色 )完美匹配,且精度達到了遠超其他模型的原子級。
在生成預測結果後,AlphaFlod3 還會提供一個置信度分數,評估該次預測結果的準確度,為研究人員提供參考。
論文展示的AlphaFlod3 的能力對於理解人類免疫反應的各個方面和新抗體的設計至關重要。這一新的工具顯然可以透過幫助研究人員了解如何接近新的疾病靶點,進而開發新的方法來追求以前遙不可及的靶點,最終加速藥物設計並提高其成功率。
此外,論文提及的RNA的預測能力同樣具備極大的想象空間。
以往的藥物靶點大部份都是蛋白質靶點,但實際上RNA會成為一個比較好的潛在靶點。透過阻斷RNA表達或阻斷RNA與蛋白質形成復合物,從而阻斷蛋白質形成功能,藥物的療效或許會比蛋白質靶點表現更好。
但在過去的采用非AlphaFlod工具進行的RNA三維結構預測中,絕大部份的預測誤差超過了10埃,與物理預測方式存在一定差距。理論上要實作RNA結構計算相關的套用,精度最好控制在2-3埃左右。
如果AlphaFlod 3能夠攻克RNA結果預測,使其預測結果達到跟蛋白質預測差不多的水平,那麽這一工具或能最佳化mRNA的蛋白表達,最佳化其穩定性,加速針對RNA target的藥物設計,甚至加速將RNA本身作為新型藥物的藥物研發。
演算法閉源,AlphaFlod 3或將開啟AI分子預測付費時代
在理想情況下,原先需要花大量時間精力和資金才能觀察到的現象,現在只需要在DeepMind的界面中輸入參數,便能在數分鐘內得到極高清晰度和準確度的生物大分子模型,甚至明確該大分子細胞系統內部的生化過程,展現如何與抗體、核酸進行反應,因而能在行業之中引起轟動。
但在實際測試中,AlphaFlod 3的能力或許不如大家期待的那樣理想。
顏寧教授團隊在微博上表示,AlphaFlod針對一個糖蛋白的預測不如上一代版本。「這次的server版本我覺得是一個速度和準確度的平衡,正確率不是最好的。我現在手上有三個都是比較奇怪的蛋白,之前我自己搭的AF2 multimer可以在很低的ranking position找到一兩個正確的conformation,這次的server版本測試全軍覆沒。」
也有學者在試用AlphaFlod 3後發現DeepMind並沒有將文章中引以為傲的蛋白-小分子配體預測任務公開,使用者仍然不能自訂配體進行復合物結構預測(aka對接)。
此外,AlphaFlod 3也因尚未開源在學界引起激烈討論。
目前,DeepMind僅為該模型釋出了一個名為AlphaFlod Server 的公共介面,該介面對可以進行實驗的分子施加了限制,僅允許每位使用者每天進行10次預測,且不提供可能與藥物結合的蛋白質結構。
在實際操作中,研究人員為獲得最高精度,需要生成大量預測結構並對其進行排名,特別是對於抗體-抗原復合物,預測質素隨著模型種子的數量增加而顯著提高,因而對工具的篩選功能提出考量。畢竟制藥公司並不關心研究人員能找出多少小分子,也不關心提供的分子是自己生成,還是從數據庫裏篩選,他們只在乎能否找到一個抑制蛋白質的最合適的小分子。
但就AlphaFlod Server現階段可以提供的服務而言,研究人員很難借助這一工具實作期望中的價值。AlphaFlod 3的使用限制中明確指出預測結果不準商用,也不能用於對接和虛擬篩選,
業內人士認為,AlphaFlod 3的開源至少會等到12月的CASP16結束後。但考慮到 Isomorphic Labs 參與了AlphaFlod 3的研發工作,DeepMInd這一次可能不會向學界開源它的開源推理程式碼或可執行檔,也不會開源演算法和原理。畢竟,這些演算法已經成為 Isomorphic Labs 的核心資產。
今年1月,Isomorphic Labs宣布與禮來和諾華達成了兩項價值30億美元的藥物發現協定,合作涉及針對多種疾病相關蛋白和途徑的治療方法的發現,正與AlphaFlod 3對抗原抗體復合體的預測能力、對蛋白配體復合體的預測能力、對蛋白核酸復合體的預測能力緊密相關。
如此來看,AlphaFlod 3的未來可能會像GPT一樣被包裝成一款商用軟件,面向不同的使用者推出不同的版本。譬如,預測結構的排序可能會成為付費專案的一部份,需要研究人員有償使用。如今絕大多數研究人員已經習慣了在論文之中附上AlphaFlod 2的預測結果,但隨著工具閉源,這一習慣或許也將逐漸改變。
不過,無論是開源還是閉源,是免費還是商用,我們都應尊重DeepMind 與 Isomorphic Labs的選擇。畢竟,面對分子生物學理解、調節生物系統復雜的原子相互作用這一命題,AlphaFlod 3確實帶領行業向前邁出了一大步,有望實作在統一的框架內準確預測各種生物分子系統的結構。
因此,合理的商用或許能夠進一步為DeepMind與 Isomorphic Labs提供更多支持,推動整個行業更快進入分子生物學的下一個時代。











