復旦大學:
新演算法突破空間組學規模化計算瓶頸

前情提要
空間組學技術在保留細胞空間位置的同時,可以精確測量分子表達資訊,為理解生理或病理狀態下的組織結構及微環境提供了前所未有的機會。空間聚類是辨識組織微環境的主要計算手段,也是目前空間組學計算領域中熱點問題。目前空間聚類演算法包括兩大類:基於圖神經網絡的演算法和基於概率圖模型的演算法。這兩大類演算法均依賴空間細胞網絡的構建,在大規模數據下,圖神經網絡存在視訊記憶體溢位問題,概率圖模型存在最佳化效率低的問題,導致目前演算法無法輕易拓展到超百萬級別數據分析。隨著多時間點、多樣本、多條件等復雜實驗設計的數據集越來越多地釋出,樣本間整合/比較分析難題也逐步顯露並亟待解決。

為此,復旦大學類腦智能科學與技術研究院原致遠建立了一種新的空間組學微環境表征和聚類演算法MENDER,解決了空間組學以上兩大難題。近日,相關成果以「MENDER: fast and scalable tissue structure identification in spatial omics data」(【MENDER:一個快速且可延伸的空間組學組織結構辨識方法】)為題,發表於Nature Communications(【自然·通訊】)期刊。
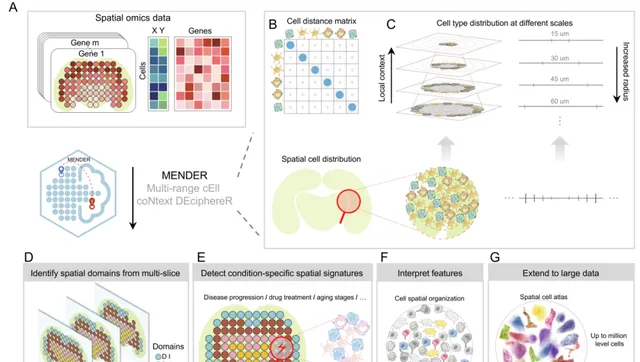
圖1 MENDER整體框架
區別於過去演算法,MENDER演算法基於多種空間組學平台之間細胞鄰域結構的保守性觀測,提出一種新型非參數化摺積核,在細胞鄰域資訊提取步驟上繞過了高時間和視訊記憶體占用的參數更新步驟,從而大振幅降低記憶體/視訊記憶體消耗,提高時間效率。在3種不同有監督任務上,效能超越目前主流圖網絡學習演算法。在無監督場景下,MENDER在3種不同組織的跨平台空間組學數據中,MENDER的效能指標均超越或與目前最優演算法持平,而時間消耗大幅減少,在25分鐘左右即可完成73萬細胞大腦空間圖譜的分析(無需GPU,Xeon(R) Gold 5218R CPU @ 2.10 GHz 80 cores)。MENDER可以在跨樣本間得到天然對齊的微環境標簽,無需額外對齊操作。
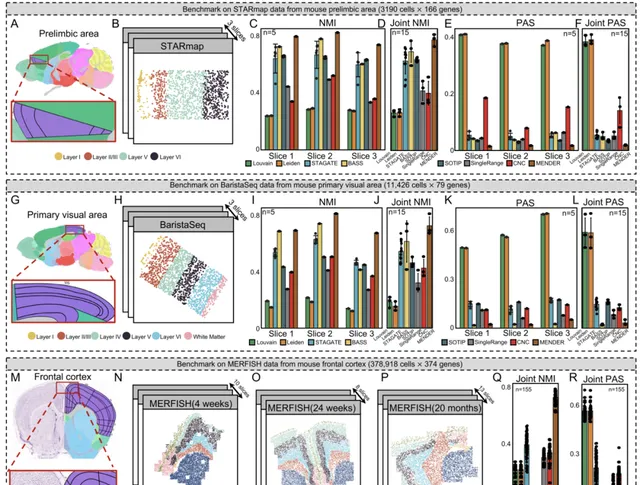
圖2 MENDER演算法效能
用MENDER分析大腦衰老空間細胞圖譜,成功辨識出兩類原文未標註出的大腦結構,其中一個大腦結構的細胞空間組成與衰老過程密切相關,只在幼年(4周齡)小鼠中出現。這一發現可以在MENDER跨切片整合和比較模組中直觀可視。MENDER提供的微環境解釋性分析也揭示了造成差異的因素主要是少突膠質細胞亞型的區域性富集。
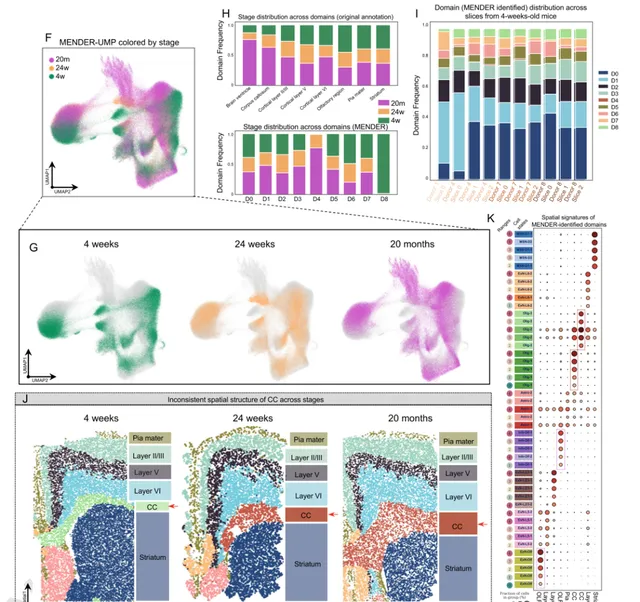
圖3 MENDER辨識衰老相關結構
最後,對一個大型三陰性乳癌病人佇列數據,采用MENDER辨識出的空間結構對病人進行表征,能成功區分開三種不同預後的病人分群,而這三種分群用傳統單細胞為單元的分析無法區分。這一例子說明了多細胞組成的空間結構或微環境對疾病發展的預測作用,同時加強了大規模數據的整合分析在空間組學大數據時代的生物學和醫學價值。
復旦大學類腦智能科學與技術研究院原致遠為本文的唯一作者。該課題得到國家自然科學基金、上海市晨光計劃、上海市啟明星專案(揚帆專項)、騰訊AI Lab犀牛鳥計劃、上海市市級科技重大專項等資助。
來源:
復旦大學官網











