
今天給大家講一篇2023年10月在arXiv上發表的 運用深度學習方法進行虛擬篩選 的文章。由於有標簽數據的稀疏性,現有深度學習的預測精度無法超過傳統對接方法。本文提出了一種新的 對比學習框架DrugCLIP ,無 需依賴於具體的親和力分數,透過對比學習方法對齊大量成對數據的結合蛋白口袋和分子的表征來根據特定蛋白檢索相應的化合物 。此外,實驗表明 DrugCLIP在不同的虛擬篩選方法上的效能顯著優於傳統的對接和有監督學習方法,有助於加速藥物發現的行程 。
虛擬篩選技術研究背景
虛擬篩選技術 在電腦輔助藥物發現(CADD)中發揮著關鍵作用,旨在 透過計算方法從龐大的化合物庫中尋找與特定蛋白質口袋相互作用的潛在藥物分子 。該方法在篩選藥物方面的有效性得益於先進的 計算能力 的崛起和大規模 生物分子數據集 的可用性,從而加速了藥物發現的行程。
過去,藥物化學家們主要關註藥物分子與靶標的結合親和力以及準確的結合位姿。然而,隨著化合物庫規模的不斷增加,這類傳統方法的計算成本和篩選效率成為制約因素。然而 虛擬篩選利用計算方法可以快速對大規模的化合物庫進行搜尋,大大提高了藥物發現的效率 。相較於實驗室篩選,成本更低,且可以在更短的時間內找到候選藥物。
近年來,專家們逐漸將虛擬篩選定義為資訊檢索任務,即 透過相似性匹配來判斷分子與給定蛋白質口袋的相關程度 ,即 從化合物庫中篩選出與目標口袋最為相似且有可能結合的分子 。相較於傳統的結合親和力預測或結合位姿的判定的方法,這一方法更註重 潛在結合分子的相似性 ,以提高篩選的精確性和效率。
模型介紹
2.1 DrugCLIP模型架構
DrugCLIP架構 主要分為兩個分支,其一為 化合物分支 ,其二為 蛋白 。編碼器是一個 SE(3) Transformer ,將每個原子的特征作為輸入。為了使得結構保持SE(3)不變性,作者引入了三維座標的幾何距離作為三維特征(圖1)。
借鑒了 語言模型bert 的思路,還引入了 隨機遮蔽原子類別型 方法進行預訓練以預測被遮蔽的原子類別型。此外,還加入 隨機雜訊 來打亂原子座標去重構原始座標。其中有一個特殊的原子分類標記[CLS]的座標位於所有原子的中心,該標記用於輸出相應蛋白質和分子的表示。損失函數的值為化合物到蛋白口袋的損失(

)加上蛋白口袋到化合物的損失(
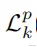
)之和的一半。公式為
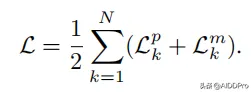
,其中N為化合物蛋白對的數量。
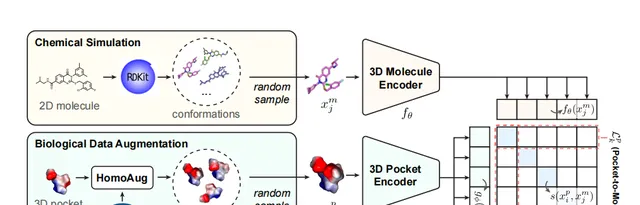
圖1DrugCLIP模型架構
2.2 數據增強方法
作者發現在蛋白口袋或分子數據中引入雜訊或擾動可能導生成化學上不合法的結構。為了解決這一問題,提出了一種新的 HomoAug增強方法 。它利用生物學中同源蛋白的概念,將PDBbind 的配體與其口袋對應的同源蛋白結合起來,將同源蛋白與配體結合,形成一個增強後的口袋配體對作為新的訓練數據(圖2)。
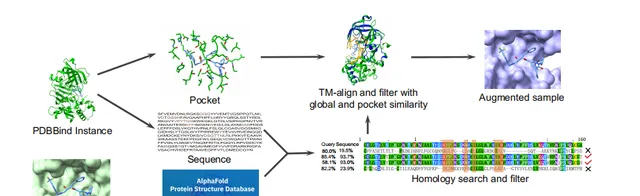
圖2 數據增強方法
實驗結果
3.1虛擬篩選效能評估
作者分析比較了兩種不同的場景:其一是 在不同化合物庫上對特定靶標進行一次性虛擬篩選 ,其二是 在特定化合物庫中對多個靶標進行多次篩選 。該實驗以機器學習打分函數(MLSF)作為基準比較。如圖3a所示,當所有的候選分子都沒有預先編碼時,該方法與其余方法篩選時間相當。然而,如果所有候選分子都進行預先編碼,DrugCLIP可以在大致30小時內對包含60億分子的Enamine數據庫進行虛擬篩選。這種時間的顯著減少表明了該方法在利用預編碼分子時的效率和可伸縮性。當搜尋庫固定時,所有的分子都預先進行編碼。在對10個靶標篩選時,DrugCLIP和MLSF之間的時間差約為10天。這項實驗突顯了當前基於機器學習的篩選方法的局限性,並表明了DrugCLIP所具有的巨大篩選效率的優勢(圖3b)。
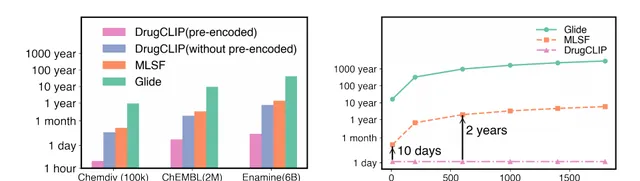
圖3 模型預測效能評估
3.2 消融實驗評估
作者對兩種不同的訓練策略進行評估:其一是 使用HomoAug方法進行數據增強 ,其二是 使用RDkit構象來取代原始的分子構象 。如圖4所示,在結合兩種不同的增強方法時,DrugCLIP模型的分類效果最佳。
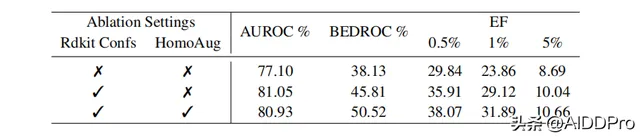
圖4消融實驗評估
結論
作者提出了一種高效、準確的 虛擬篩選的新方法(DrugCLIP) 。該方法 利用對比學習方法來對齊繫結口袋和分子的表示 。在不同的虛擬篩選任務中該方法均有競爭性的優勢,不僅提高了篩選準確性,而且顯著縮短了大規模虛擬篩選所需的時間。這為從數十億種化合物的虛擬庫篩選候選藥提供了可能性。此外, 透過不斷著眼於探索新的數據增強方法及原子級相互作用將助於更有效地發現潛在的候選藥物 。
參考文獻
- Michael Brocidiacono, Paul Francoeur, Rishal Aggarwal, Konstantin Popov, David Koes, and Alexander Tropsha. Bigbind: Learning from nonstructural data for structure-based virtual screening. 2022
版權資訊
本文系AIDD Pro接受的外部投稿,文中所述觀點僅代表作者本人觀點,不代表AIDD Pro平台,如您發現釋出內容有任何版權侵擾或者其他資訊錯誤解讀,請及時聯系AIDD Pro (請添加微訊號sixiali_fox59)進行刪改處理。
本文為原創內容,未經授權禁止轉載,授權後轉載亦需註明出處。有問題可發郵件至[email protected]











