染色質免疫沈澱結合測序(ChIP-seq)是一種強大的工具,用於研究體內DNA與組織蛋白的交互作用。盡管ChIP-seq已被廣泛使用,但在植物中套用仍然具有挑戰性,尤其是當樣本富含初級或次級代謝物(如種子)或起始材料有限時。傳統的ChIP實驗由於對大量植物材料的需求以及眾多勞動密集型步驟,顯著限制了實驗的規模,這往往對缺乏ChIP經驗的實驗室構成障礙。
2024年9月15日, New Phytologist 線上發表了題為 "A simple, robust, cost-effective, and low-input ChIP-seq method for profiling histone modifications and Pol II in plants" 的研究論文。本文發了一種新方法—UP-ChIP(Ultrasensitive Plant Chip-seq),該方法能夠減少實驗的勞動強度、成本和所需樣本量,並且適用於植物中組織蛋白修飾和RNA聚合酶II(Pol II)的分析。研究建議使用UP-ChIP替代傳統ChIP-seq,尤其是在植物樣本中分析組織蛋白修飾和Pol II時的起始材料數量有限時。

UP-ChIP方法結合了超低輸入的天然ChIP-seq和ChIPmentation的優點,適用於處理多種植物樣本(如純化的細胞核、新鮮幼苗和種子)。其流程(圖1)包括使用液氮將樣本研磨成粉末,隨後透過微球菌核酸酶(MNase)處理釋放染色質,一部份碎片作為輸入DNA,其余染色質則透過抗體包被的磁珠進行免疫沈澱。沈澱的DNA和輸入樣本經過Tn5轉座酶反應直接添加測序接頭,純化後透過PCR擴增構建文庫,並在Illumina平台上進行測序。整個過程可以在2天內完成,並具有高靈敏度、低試劑成本和低抗體需求,相較於傳統ChIP方法具有顯著優勢。
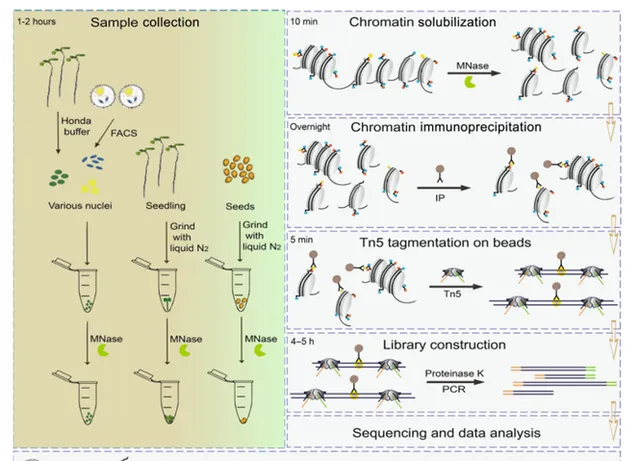
圖1 UP-ChIP方法的工作流程
研究中展示了透過UP-ChIP方法在不同品質的阿拉伯芥幼苗細胞核樣本(20 mg、10 mg、5 mg和2.5 mg)中對pK27me3組織蛋白修飾進行分析的結果(圖2)。實驗表明,透過Honda緩沖液分離的細胞核樣本非常適合UP-ChIP方法。數據品質高,表現為明顯的峰值與背景訊號差異、大量重疊的峰值、以及符合預期的pK27me3訊號覆蓋整個基因的模式。即使使用最少量的2.5 mg細胞核樣本,實驗依然產生了良好的結果,表明UP-ChIP方法對低輸入樣本具有良好的適用性,並且可能在更低的起始材料下也能獲得可接受的結果。
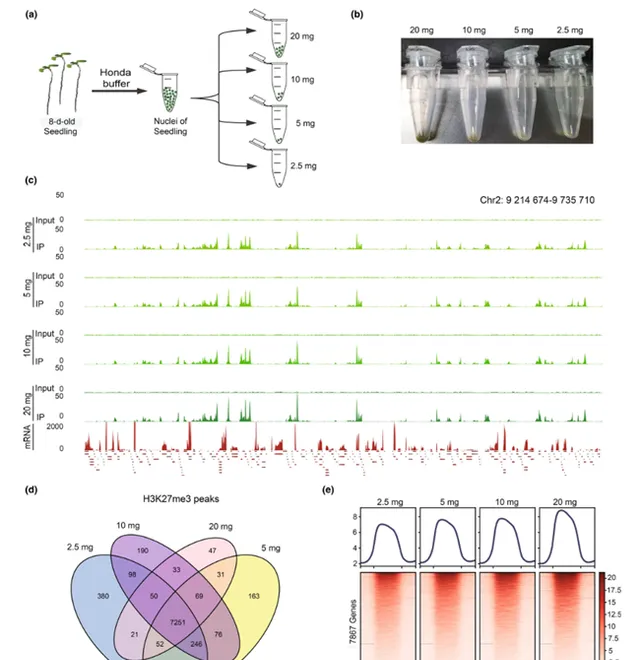
圖2 UP-ChIP在不同品質的幼苗核上的靈敏度和可靠性測試
該實驗還考慮了簡化UP-ChIP流程的可能性,透過直接使用新鮮植物組織而跳過細胞核提取步驟。研究人員對單個阿拉伯芥幼苗及不同數量的種子(20、30、40、50顆)進行了pK27me3 UP-ChIP分析(圖3)。結果表明,UP-ChIP在未提取細胞核的情況下,依然能夠獲得與傳統ChIP方法相當的結果,且只需使用少量樣本,如單個幼苗或2.5 mg細胞核。對種子的UP-ChIP雖然挑戰更大,但透過添加氯仿處理染色質以去除脂肪酸,成功解決了脂肪酸和多糖含量高的問題。實驗還發現,透過增加種子數量,可以提高測序比對率。MNase濃度的實驗進一步表明,適量的MNase(1-2 U/µl)能產生最佳的ChIP結果,而過高的濃度則會影響訊號的連續性和樣本的重復性。總體而言,UP-ChIP在植物種子和幼苗中表現出良好的適用性,但對於富含澱粉的種子(如水稻)可能需要進一步最佳化。
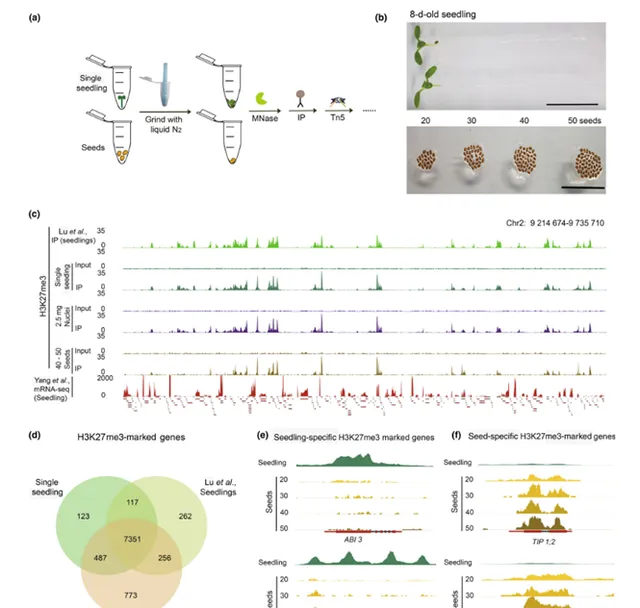
圖3 單個幼苗或種子樣品的UP-ChIP測試
此外,本研究還展示了UP-ChIP方法在阿拉伯芥幼苗中用於分析RNA聚合酶II(Pol II)結合模式的結果(圖4)。與大多數非組織蛋白染色質蛋白不同,Pol II在MNase消化中與DNA形成穩定復合體,能夠抵抗消化。在實驗中,使用針對Pol II Ser2P CTD的抗體進行了UP-ChIP,結果檢測到大量Pol II峰,尤其集中在活躍轉錄的基因3'末端。UP-ChIP獲得的Pol II結合圖譜與pNET-seq方法的結果高度一致。進一步分析還表明,Pol II和pK27me3的訊號呈現相互排斥的模式,驗證了UP-ChIP在Pol II分析中的可靠性。總的來說,UP-ChIP可以在不進行化學固定和細胞核提取的情況下,成功分析基因組範圍內的組織蛋白修飾和Pol II結合,簡化了實驗步驟。
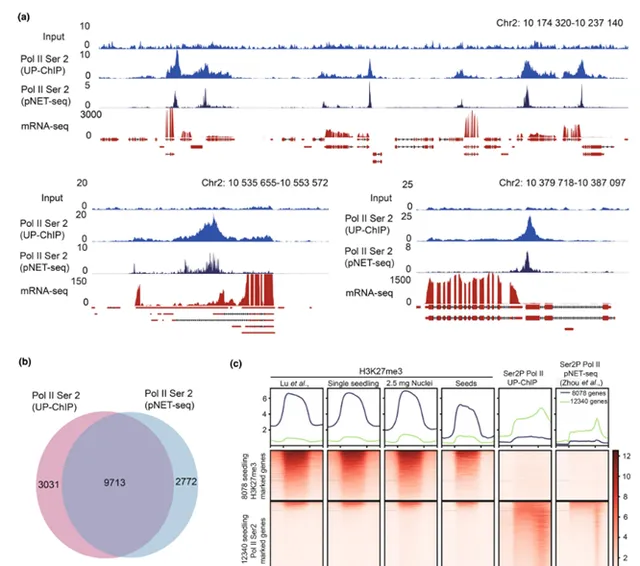
圖4 UP-ChIP在阿拉伯芥幼苗中進行Pol II分析的測試
最後,研究中人員開展了UP-ChIP方法在檢測阿拉伯芥pK27me3缺失突變體(swn clf)的可行性實驗(圖5)。PRC2復合體的關鍵亞基CLF和SWN負責在植物營養組織中沈積pK27me3,抑制基因表現。突變體swn clf中pK27me3的缺失導致幼苗發育缺陷。UP-ChIP對該突變體的分析顯示,僅產生背景訊號,表明UP-ChIP能夠準確反映pK27me3的缺失。進一步的實驗透過混合不同比例的野生型Col-0和swn clf萃取物,驗證了UP-ChIP對組織蛋白修飾水平變化的敏感性,結果顯示隨著swn clf比例的增加,pK27me3訊號逐漸減弱。盡管UP-ChIP對組織蛋白修飾變化具有靈敏性,但與其他ChIP-seq方法類似,精確量化仍具有挑戰性。
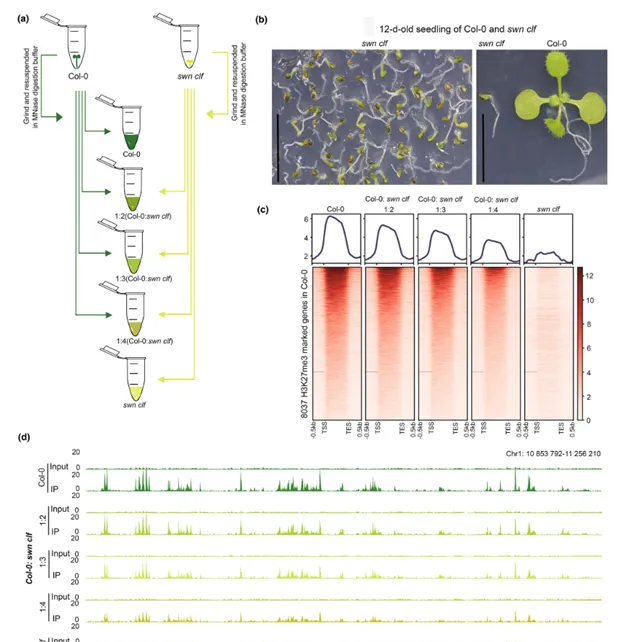
圖5 對具有pK27me3的幼苗樣本進行UP-ChIP檢測
總的而言,UP-ChIP方法是傳統ChIP-seq的替代方案,特別適用於樣本量有限的情況。它簡化了實驗流程,省去了繁瑣的步驟,同時降低了成本。然而,該方法可能不適用於分析瞬時結合的蛋白質,也難以進行單細胞分析。但是,對於沒有豐富資源或ChIP-seq經驗的實驗室來說,UP-ChIP提供了一種低成本且高效的解決方案,能夠在低輸入的植物樣本中分析表觀遺傳修飾。這將使得更多的植物遺傳學和表觀遺傳學研究得以實作。
原文連結:
https://doi.org/10.1111/nph.20125
小麥族多組學網站: http://wheatomics.sdau.edu.cn
投稿、合作等信箱: [email protected]












